D.2 Contenu
Le rapport sert à restituer de façon synthétique les résultats d’une étude scientifique, et les interprétations. Le tout est remis dans le contexte de la bibliographie existante en la synthétisant dans l’introduction et en comparant les résultats avec d’autres études connexes dans la discussion. Il faut garder à l’esprit qu’un lecteur doit comprendre l’intégralité du rapport avec un minimum de connaissances a priori sur l’étude réalisée, mais avec des connaissances générales dans la spécialité. Donc, un rapport sur un sujet biologique est adressé à un lecteur biologiste pour lequel il ne faut pas rappeler les concepts de base dans sa discipline. Par contre, il faut expliquer avec suffisamment de détails comment l’étude a été réalisée dans la section “matériel et méthodes”.
En général, les phrases sont simples, directes, courtes et précises (veuillez à utiliser le vocabulaire adéquat et précis). Les explications sont, autant que possible, linéaires. Evitez les renvois dans différentes autres parties du rapport, si ce n’est pour rappeler un élément évoqué plus haut, ou pour se référer à une figure ou une table. A ce sujet, les figures (dont les images, photos, schémas et graphiques) sont numérotées (Figure 1, Figure 2, …) et accompagnées d’une légende en dessous d’elles. La figure et sa légende doivent être compréhensibles telles quelles. Dans le texte, vous pourrez alors vous référer à la figure, par exemple: “Tel phénomène est observable (voir Fig. 3)”, ou “La Fig. 4 montre …”. idem pour les tableaux qui sont également numérotés (Tableau 1, Tableau 2, …) et légendés, mais au dessus du tableau. Les règles de lisibilité du tableau + légende et de renvoi vers les tableaux sont identiques que pour les figures. Les équations peuvent aussi être numérotées et des renvois de type (eq. 5) peuvent être alors utilisés. Enfin, toute affirmation doit être soit démontrée dans le rapport, soit complétée d’une citation vers un autre document scientifique qui la démontre. La partie bibliographie regroupe la liste de tous les documents qui sont ainsi cités à la fin du rapport.
Veuillez à respecter les notations propres au système métrique international, les abbrévations usuelles dans la discipline, et le droit d’auteur et les licenses si vous voulez citer un passage ou reprendre une illustration provenant d’un autre auteur (sans omettre d’indiquer qui en est l’auteur). Enfin, en vue de rendre le document parfaitement reproductible, vous pouvez indiquer dans les annexes où trouver la source (le document .Rmd) et les données analysées. Vous pouvez également terminer avec un chunk qui renseigne de l’état du système R utilisé, y compris l’ensemble des packages employés. Ce chunk, présenté en annexe, contiendra l’instruction utils::sessionInfo(), ou mieux : sessioninfo::session_info() (version courte) ou devtools::session_info() (version longue). Par exemple :
sessioninfo::session_info()─ Session info ──────────────────────────────────────────────────────────
setting value
version R version 3.4.4 (2018-03-15)
os Ubuntu 16.04.5 LTS
system x86_64, linux-gnu
ui X11
language (EN)
collate en_US.UTF-8
tz Europe/Brussels
date 2020-08-21
─ Packages ──────────────────────────────────────────────────────────────
package * version date
acepack 1.4.1 2016-10-29
assertthat 0.2.0 2017-04-11
backports 1.1.2 2017-12-13
base64enc 0.1-3 2015-07-28
bindr 0.1.1 2018-03-13
bindrcpp 0.2.2 2018-03-29
bookdown 0.7 2018-02-18
broom 0.4.4 2018-03-29
cellranger 1.1.0 2016-07-27
chart * 1.2.0 2018-09-16
checkmate 1.8.5 2017-10-24
cli 1.0.0 2017-11-05
clisymbols 1.2.0 2017-05-21
cluster 2.0.7-1 2018-04-09
codetools 0.2-15 2016-10-05
colorspace 1.3-2 2016-12-14
cowplot 0.9.2 2017-12-17
crayon 1.3.4 2017-09-16
data.io * 1.2.1 2018-10-27
data.table 1.10.4-3 2017-10-27
digest 0.6.15 2018-01-28
dplyr * 0.7.4 2017-09-28
ellipse 0.4.1 2018-01-05
evaluate 0.10.1 2017-06-24
flow * 1.1.0 2018-08-29
forcats * 0.3.0 2018-02-19
foreign 0.8-69 2017-06-21
Formula 1.2-2 2017-07-10
ggplot2 * 2.2.1 2016-12-30
ggplotify 0.0.3 2018-08-29
ggpubr 0.1.6 2017-11-14
glue 1.3.0 2018-08-29
gridExtra 2.3 2017-09-09
gridGraphics 0.3-0 2018-04-03
gtable 0.2.0 2016-02-26
haven 1.1.1 2018-01-18
Hmisc 4.1-1 2018-01-03
hms 0.4.2 2018-03-10
htmlTable 1.11.2 2018-01-20
htmltools 0.3.6 2017-04-28
htmlwidgets 1.2 2018-04-19
httr 1.3.1 2017-08-20
igraph 1.2.1 2018-03-10
inline 0.3.14 2015-04-13
jsonlite 1.5 2017-06-01
knitr 1.20 2018-02-20
lattice * 0.20-35 2017-03-25
latticeExtra 0.6-28 2016-02-09
lazyeval 0.2.1 2017-10-29
lubridate 1.7.4 2018-04-11
magrittr 1.5 2014-11-22
MASS * 7.3-49 2018-02-23
Matrix 1.2-14 2018-04-09
mnormt 1.5-5 2016-10-15
modelr 0.1.1 2017-07-24
munsell 0.4.3 2016-02-13
nlme 3.1-137 2018-04-07
nnet 7.3-12 2016-02-02
pillar 1.2.1 2018-02-27
pkgconfig 2.0.1 2017-03-21
plyr 1.8.4 2016-06-08
proto 1.0.0 2016-10-29
pryr 0.1.4 2018-02-18
psych 1.8.3.3 2018-03-30
purrr * 0.2.4 2017-10-18
R6 2.2.2 2017-06-17
RColorBrewer 1.1-2 2014-12-07
Rcpp 0.12.16 2018-03-13
readr * 1.1.1 2017-05-16
readxl 1.0.0 2017-04-18
reshape2 1.4.3 2017-12-11
rlang 0.2.0 2018-02-20
rmarkdown 1.10 2018-06-11
rpart 4.1-13 2018-02-23
rprojroot 1.3-2 2018-01-03
rstudioapi 0.7 2017-09-07
rvcheck 0.0.9 2017-07-10
rvest 0.3.2 2016-06-17
scales 0.5.0 2017-08-24
SciViews * 1.1.0 2018-09-28
sessioninfo 1.0.0 2017-06-21
stringi 1.1.7 2018-03-12
stringr * 1.3.0 2018-02-19
survival 2.42-3 2018-04-16
svMisc * 1.1.0 2018-08-29
tibble * 1.4.2 2018-01-22
tidyr * 0.8.0 2018-01-29
tidyverse * 1.2.1 2017-11-14
tsibble 0.1.5 2018-04-01
viridis 0.5.1 2018-03-29
viridisLite 0.3.0 2018-02-01
withr 2.1.2 2018-03-15
xfun 0.1 2018-01-22
xml2 1.2.0 2018-01-24
yaml 2.1.18 2018-03-08
source
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
Github (SciViews/chart@26696d4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
Github (SciViews/data.io@b9a69c1)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
Github (SciViews/flow@8fb8690)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
Github (GuangchuangYu/ggplotify@a6fd39a)
CRAN (R 3.4.4)
Github (tidyverse/glue@4e74901)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
Github (SciViews/SciViews@8808306)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
Github (SciViews/svMisc@c61c525)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4)
CRAN (R 3.4.4) D.2.1 Table des matières
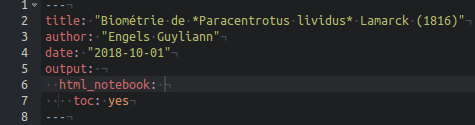
La table des matières est d’une importance capitale pour un long document (mais facultative pour un plus court rapport) afin de présenter la structure de votre oeuvre aux lecteurs. Heureusement, il n’est pas nécessaire de l’écrire manuellement. La table des matières est générée automatiquement dans un rapport R Markdown. L’instruction à ajouter dans le préambule du document R Notebook afin d’obtenir une table des matières est toc: yes (ne l’encodez pas directement, mais sélectionnez l’option Include table of contents dans les options de formattage du document accessibles à partir du bouton engrenage à droite de Preview ou Knit -> Output Options...). Lorsque vous fermerez cette boite de dialogue de configuration, l’entrée ad hoc sera ajoutée pour vous dans le préambule.
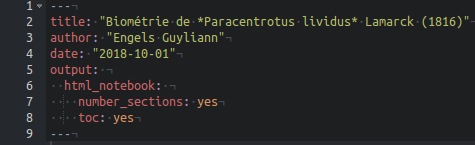
Vous pouvez aussi choisir de numéroter vos titres automatiquement. L’instruction à ajouter en plus de toc: yes dans le préambule du document R Notebook afin d’obtenir une table des matières avec des titres numéroté est number_sections: yes. Encore une fois, passez par la boite de dialogue de configuration, et cochez-y l’entrée Number section headings.
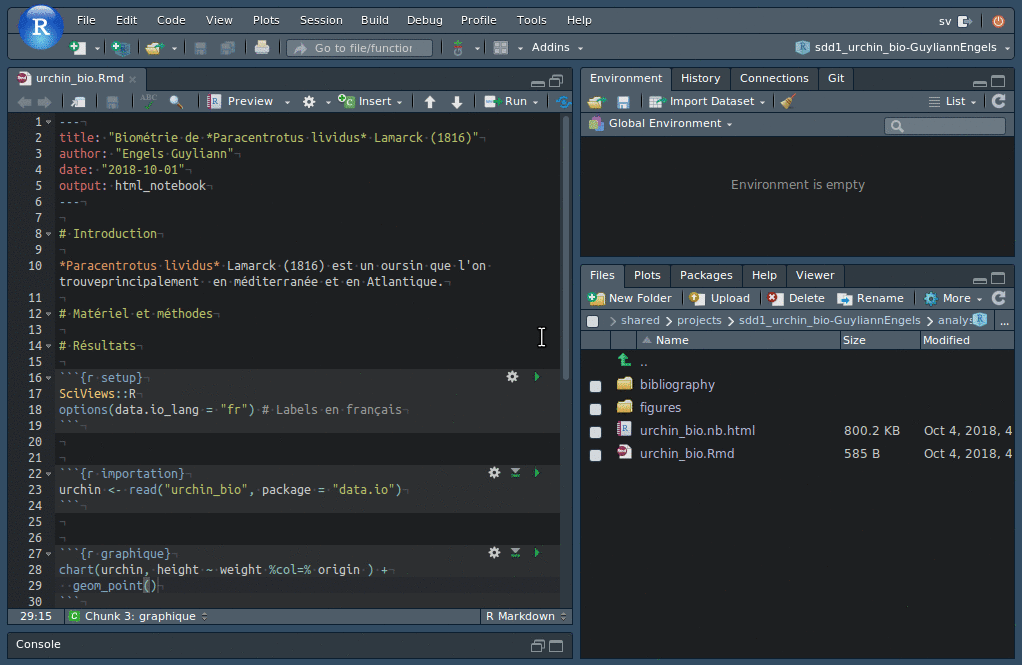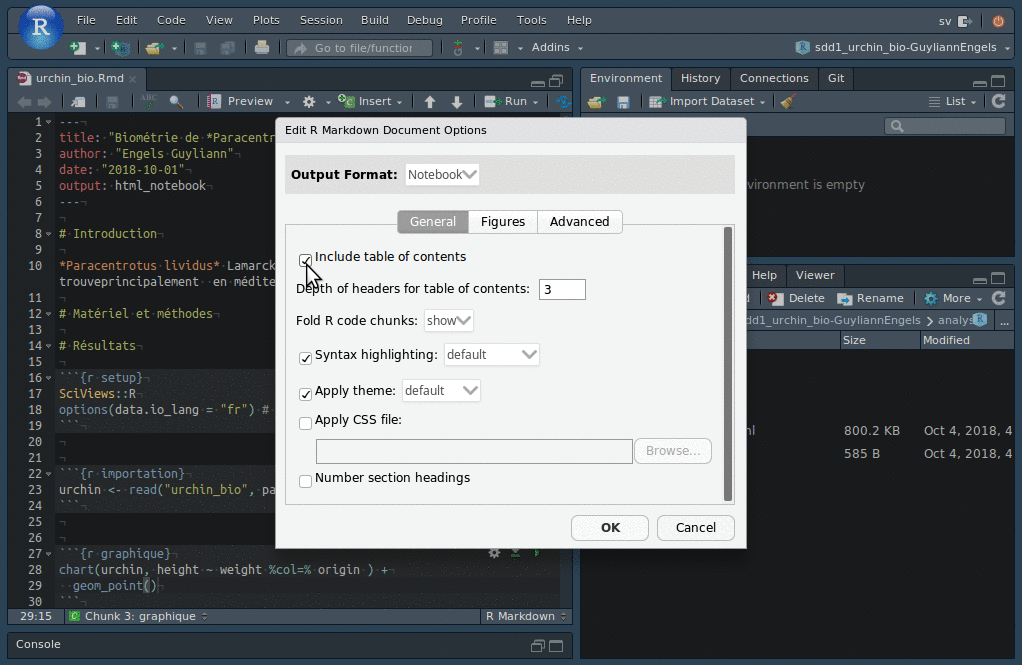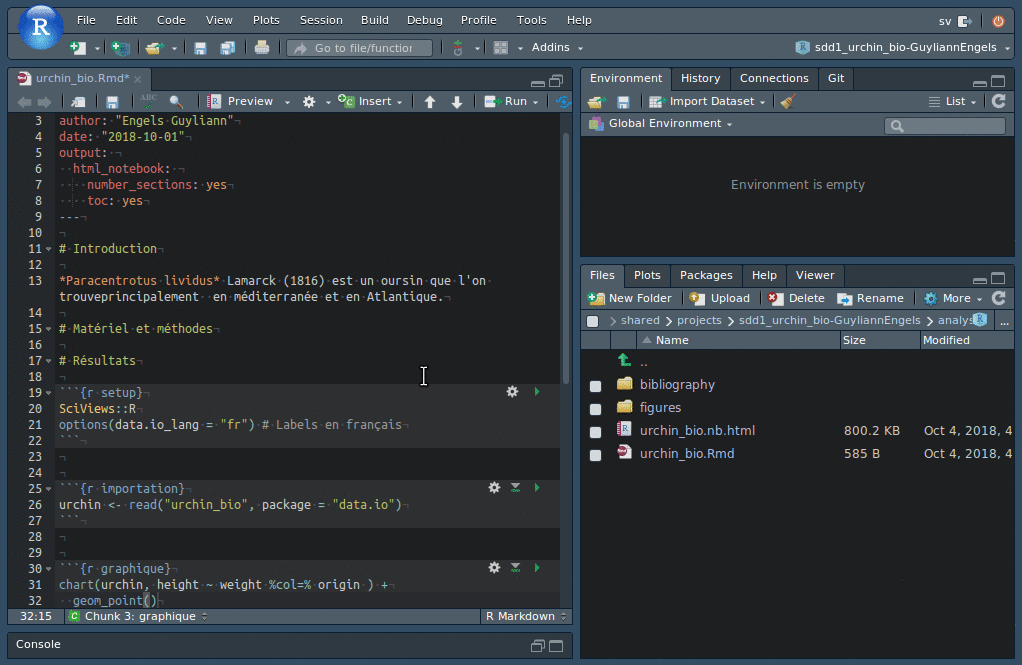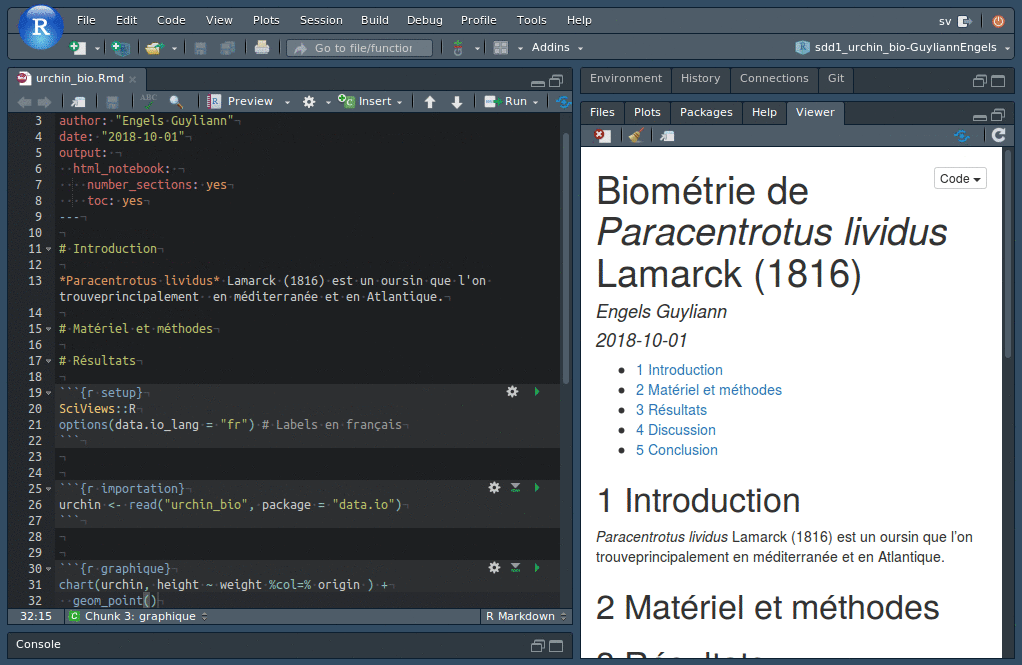
Voyez l’animation ci-dessous pour accéder à la boite de dialogue de configuration du document R Markdown/R Notebook.
D.2.2 Introduction
L’introduction d’un rapport (ou d’un mémoire) a pour principal objectif de replacer l’étude scientifique réalisée dans son contexte. La règle la plus importante est qu’un lecteur n’ayant jamais entendu parler de cette étude doit comprendre l’intégralité du rapport. L’introduction doit donc permettre de :
- Remettre l’expérience dans son contexte,
- Décrire l’organisme étudié
- caractéristiques générales de l’organisme, distribution géographique, biotope,…
Notez que l’ajout d’images ou d’une carte de distribution est un plus dans l’introduction.
D.2.3 But
Le but permet de synthétiser la question posée dans cette étude en fonction du contexte de l’expérience expliqué dans l’introduction.
D.2.4 Matériel & méthodes
La section matériel & méthodes permet de décrire les aspects techniques de l’étude comme le matériel employé et les méthodes mises en oeuvre (protocoles des manipulations et des mesures effectuées) afin d’acquérir les données. Cette section est également le lieu de description des techniques statistiques utilisées pour analyser les données, des programmes informatiques employés, …
D.2.5 Résultats
Les résultats vont généralement contenir deux parties :
- La description des données, via l’exploration des données récoltées (avec graphiques et/ou estimateurs statistiques)
- L’application des outils statistiques pertinents pour répondre à la question posée
D.2.6 Discussion
Cette section comprend l’interprétation biologique des résultats et la remise dans un contexte plus général, notamment en les comparant à des observations connexes réalisées par d’autres auteurs scientifiques. Il est d’une importance capitale d’avoir un regard critique sur les résultats obtenus. Cette mise en contexte aide en ce sens.
D.2.7 Conclusion(s)
Cette section va résumer les principales implications à retenir de notre étude et, éventuellement, proposer des perspectives afin de poursuivre la recherche dans cette thématique.
D.2.8 Bibliographie (ou références)
La rédaction de travaux s’appuye toujours sur une recherche bibliographique au préalable. CIl faut documenter convenablement les sources bibliographiques au sein de cette section afin d’éviter le plagiat volontaire ou involontaire. Une multitude de programmes existent pour faciliter la gestion de votre base de données bibliographique comme Mendeley, Zotero ou encore Endnote.
- Pour générer correctement ses références bibliographiques dans un document R Markdown/R Notebook, consulter ceci. Il s’agit d’un manuel en anglais de RStudio qui explique comment faire dans le détail.